関連記事
東大、細胞内ビッグデータから多階層ネットワークを再構築する手法を開発
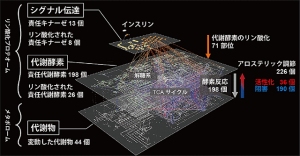
リン酸化プロテオーム階層とメタボローム階層をつなぐことにより得られた、インスリン代謝制御ネットワークを示す図。変動した代謝物44個の制御因子として代謝酵素198個、責任キナーゼ13個、アロステリック調節226個が関与する大規模ネットワークとなっている(東京大学の発表資料より)[写真拡大]
東京大学の柚木克之助教らによる研究グループは、2つのオミクス階層(生体内の物性がよく似た分子同士を集めたグループ)にまたがるネットワークを再構築する手法を開発し、インスリンが作用する分子のネットワークの全貌を初めて明らかにした。
細胞は、DNA、RNA、タンパク質、代謝物質などからできており、物性がよく似た分子同士のグループをオミクス階層と言う。これまで、異なるオミクス階層間を繋ぐネットワークは明らかになっていなかった。
今回の研究では、細胞内のビッグデータから大規模な多階層ネットワークを自動的に再構築する手法「トランスオミクス解析」を開発し、インスリンの投与によって生じる経時的な変化に適用したところ、インスリンが作用する分子のネットワークの全貌が明らかとなった。
このインスリン代謝制御ネットワークを細かく解析することにより、血糖値の新規な調節メカニズムが解明できる可能性がある。また、「トランスオミクス解析」はインスリン以外にも、幅広い生命機能の背後にある多階層にまたがる分子ネットワークの解明に応用でき、生命機能や疾患のメカニズム解明や制御につながることが期待される。
スポンサードリンク
