関連記事
名大、100万原子を超えるフラーレン群の構造解析に成功
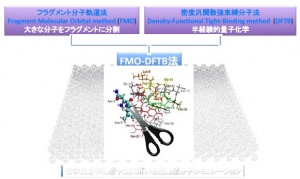
名古屋大学の研究グループが開発したFMO-DFTB法のイメージ(名古屋大学の発表資料より)[写真拡大]
名古屋大学のステファン・イレ教授と西本佳央大学院生らによる研究グループは、100万原子以上を持つ分子を高速で解析する新しいコンピューターシミュレーション法を開発した。
生化学やナノエレクトロニクスの分野では、タンパク質やフラーレン群など1万~100万個の原子からなる分子も多く、これまでは解析に膨大な時間とコンピューターメモリが必要であった。
今回の研究では、フラグメント分子軌道法と密度汎関数強束縛分子法を組み合わせたFMO-DFTBという新しいコンピューターシミュレーション法を開発した。その結果、100万原子を超えるフラーレン群などの構造解析に成功し、約2万個の原子から成る水分子群を解析した際にはこれまでの約1万倍の速度(3週間から3分に短縮)で解析結果を得ることができた。
今後は、化学反応、生体分子やナノエレクトロニクス材料の構造解析など、様々な研究分野で応用可能であると期待されている。
なお、この内容は「Journal of Chemical Theory and Computation」オンライン版に掲載された。
スポンサードリンク
