関連記事
ゲノム情報の「見える化」、AIが効率的に解析 理研などが新手法開発
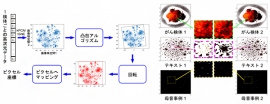
ゲノム情報等の非画像データを画像化する方法(左)と実例(右)(写真:理研の発表資料より)[写真拡大]
約30億個存在するとされるヒトゲノム。これらゲノムや遺伝子情報を活用することで、医療診断など幅広い活用が可能になる。理化学研究所(理研)は6日、深層学習(ディープラーニング)により、ゲノムや遺伝子情報等の非画像データを画像データに変換する方法を開発したと発表した。
【こちらも】理研、ゲノム解析で日本人特有の遺伝的変異を解明 飲酒量や血糖値等に関係
■解析が困難なゲノム情報
ゲノムや遺伝子情報は、病気の個人差解析や診断に役立つ。ところがこれらの情報は、数万から数千万もの変数をもつ超高次元データであるため、伝統的な統計学的手法では解析が困難だという。
理研の研究者らで構成される国際グループが着目したのが、ディープラーニングなどの機械学習である。機械学習は多くの変数から重要な変数群を抽出でき、新規のサンプルが特定の病気と関連あるかを判定するにも応用できる。しかし膨大なゲノム情報は背後に複雑な構造があるため、適切に変数を配置する等の事前処理が必要になる。
研究グループが今回開発したのが、「ディープインサイト法」と呼ばれる、ゲノム情報の事前処理から、特徴抽出や分類まで実行する手法である。
■「見える化」することでAIが適切に解析
ディープインサイト法ではまず、類似した変数や生データがクラスタとして変換される。似た変数をまとめて配置し、異なるものを離して配置することで、変数が画素として扱われるという。
作成された画像解析に役立つのが、深層学習のひとつである畳み込みニューラルネットワーク(CNN)である。CNNは画像内の構造を捉え、特徴を抽出し画像を分類するのに適している。
研究グループはディープインサイト法の検証のために、遺伝子発現データ、テキストデータ、母音データ等の多様なデータを活用し、従来の人工知能手法である決定木やランダムフォレスト法等の手法と結果を比較した。その結果、平均分類精度が95%と高精度であると判明。この数値は、従来手法で最高の結果を出したランダムフォレスト法の86%を大幅に更新する。
研究グループによると、ディープインサイト法によりDNA配列やタンパク質配列等のデータを解析できるという。今後は、同手法により医学・生命科学の複雑なデータの解析が進み、個人ごとの診断や予測に役立てられるだろうと期待を寄せている。
研究の詳細は、英オンライン科学誌Scientific Reportにて6日に掲載されている。(記事:角野未智・記事一覧を見る)
スポンサードリンク
スポンサードリンク
- Google、超リアルな動画生成AI「Lumiere」を発表 1/31 15:40
- AmazonのFire TVシリーズ、画像生成AIによる壁紙機能が追加 1/26 09:13
- Chromeに新たなAI機能、タブの自動整理や文章作成支援など 1/26 09:10
- Fairly Trained始動、学習データを無断使用しない生成AIに認定証を授与 1/21 17:37
- 生成AIが書いた論文を瞬時に判定する「生成AIチェッカー」、ユーザーローカル 1/19 16:39
